当前探测水溶液中蛋白质结构的动态变化仍是一个巨大挑战。针对这个问题,日前我组提出了一种基于质谱分析的尺寸选择VAILase酶切(SVP)的新策略。
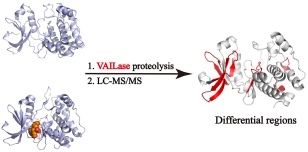
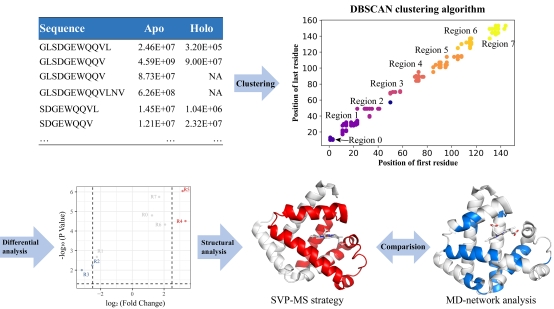
目前结构蛋白质组学研究中,活性蛋白质限制性酶切(LiP)-质谱方法在酶切过程中需要对蛋白酶用量和酶切时间进行严格的限制,通过酶切运动学(kinetics)控制实现酶切对蛋白质结构的选择性。本论文工作利用VAILase六聚体笼状结构中底物进入孔洞(3 nm)的尺寸选择性以及笼状结构中底物识别结构域和酶切活性位点之间的六把“分子尺”,实现了对活性蛋白质在正常酶用量和酶切时间下的高度结构选择性,并将其命名为尺寸选择VAILase酶切(SVP)。本工作结合SVP策略和分子动力学模拟(molecular dynamics simulation),成功探测了holoMb-apoMb的结构转换过程以及不同外源小分子抑制剂诱导Aurora A激酶的细微构象变化的动态细节,与分子动力学模拟结果高度吻合。我们的工作为探索水溶液中蛋白质结构与功能的关系提供了新思路。
相关成果以“Size-Selective VAILase Proteolysis Provides Dynamic Insights into Protein Structures”为题,于近日发表在《分析化学》(Analytical Chemistry)上。该工作的并列第一作者为我组2021届博士孙彬文和研究助理吕骥,通讯作者为王方军研究员。

